Autoencoders Variazionali e Bioinformatica
Introduzione agli autoencoder variazionali e alcune loro possibili applicazioni ad analisi di bioinformatica/metagenomica
Articolo in lingua originale in lingua inglese di Anuradha Wickramarachchi
In generale, gli autoencoder mirano ad imparare una rappresentazione dei dati in una dimensione inferiore rispetto a quella di partenza.Uno dei maggiori vantaggi degli autoencoder è quello di essere in grado di imparare dimensioni significativamente piccole al contrario delle decomposizioni simili alla PCA che sono limitate dalla loro natura lineare. Sentitevi liberi di dare un’occhiata anche il mio articolo riguardo gli autoencoders.

Come al solito, parlerò di un’applicazione nell’ambito della bioinformatica
Autoencoders Variazionali
Rispetto agli autoencoder tradizionali (AEs), quelli variazionali (VAEs) appartengono alla famiglia dei modelli generativi. Questo perché i VAEs imparano distribuzioni latenti dei dati in input. Dai nuovi punti appartenenti a queste distribuzioni, gli autoencoder variazionali sono in grado di costruire nuovi punti di dati. Tuttavia, l’aspetto generativo dei VAEs non viene utilizzato spesso perché per tale compito le GAN (generative Adversarial Networks) sono comunque più performanti.
Una caratteristica di significativa importanza dei VAEs è l’abilità di imparare diverse distribuzioni dei dati (similmente alle miscele Gaussiane). Queste distribuzioni aiutano a raggruppare i dati che presentano distribuzioni sottostanti diverse.
Import necessari in Python
Per far funzionare i prossimi script di codice è necessario importare le seguenti librerie:
import torch from torch.nn import functional as F from torch import nn, optim from torch.utils.data import DataLoader from torch.utils.data.dataset import TensorDataset import numpy as np from tqdm import tqdm, trange import seaborn as sns import umap import matplotlib.pyplot as plt
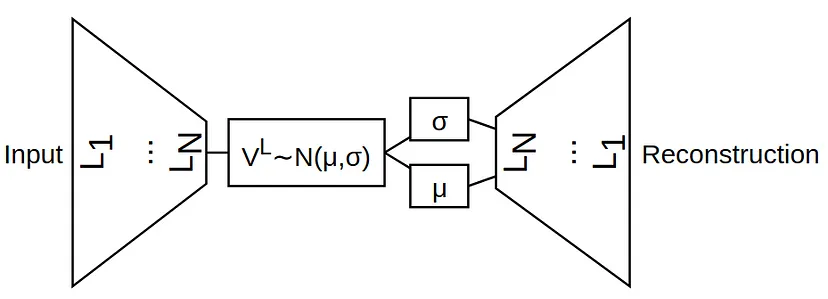
L’archietettura di un VAE
Simile ad un AE, c’è una regione a collo di bottiglia seguita da una ricostruzione. Più formalmente, abbiamo un encoder e un decoder. Da notare la rappresentazione latente VL e le successive variabili mu e sigma. La ricostruzione è generata dalle distribuzioni di questi parametri. La classe completa del VAE viene rappresentata come segue:

Trucco di ri-parametrizzazione
Da notare che, abbiamo le variabili sigma e mu da passare nel gradiente. In altre parole, dovremmo impostare il VAE in modo che impari adeguatamente sigma e mu dato un dataset in ingresso. Questo risultato si ottiene utilizzando il trucco di parametrizzazione, ovvero:
decoder_input = mu + epsilon * e ^ std
Si noti che, nel momento in cui viene scelto e^std, quello che si ottiene è una varianza logaritmica. Per questo nel codice si fa riferimento a logvar.
Funzioni di perdita e di attivazione
Si noti che viene utilizzata la funzione di attivazione RELU in molte occasioni. Tuttavia, nel caso della variabile latente logvar, viene scelto softplus. Questo perchè la varianza logaritmica è sempre positiva. Nella ricostruzione finale si utilizza una sigmoide dal momento che le dimensioni dei dati in input variano tra 0 e 1.
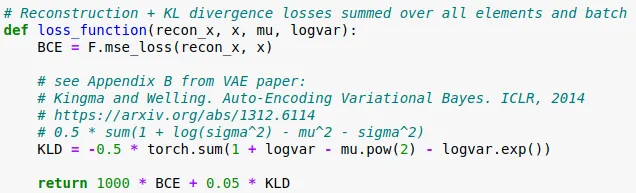
La funzione di perdita è costituita da due parti. La ricostruzione della perdita (RL) e la divergenza-KL (KLD). In particolare si utilizza KLD per assicurare che le distribuzioni apprese dalla rete siano il più vicine possibile ad una distribuzione normale (o Gaussiana, o similari). Potete leggere di più qui.
La ricostruzione è un buon vecchio Errore Quadratico Medio. In base all’applicazione, però, questo potrebbe cambiare. Ad esempio, per immagini in bianco e nero (MNIST) si userebbe la Binary Crossentropy.
Infine, possiamo cercare iperparametri ad-hoc per i pesi della RL e della KLD in modo che permettano il miglior clustering (binning o altro).
Eseguire esempi con PyTorch
Consideriamo ora il seguente dataset di metagenomica da uno dei miei paper più recenti. Le reads più lunghe sono state simulate utilizzando l’applicazione SimLoRD.
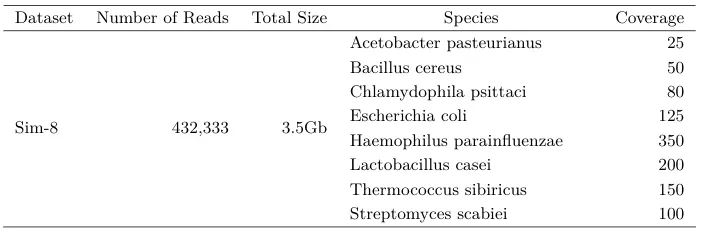
Ora il dataset è stato vettorizzato utilizzando il tool: https://github.com/anuradhawick/seq2vec, inteso
per la generazione dei dati nel Machine Learning in bioinformatica.
Il caricamento dei dati può essere realizzato come segue:

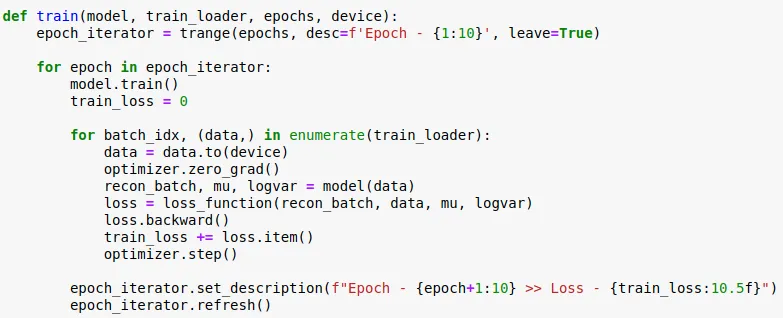
Mentre la funzione per addestrare il VAE:
L’inizializzazione
data = LOAD DATA truth = LOAD GROUND TRUTH # for visualizationdevice = "cuda" if torch.cuda.is_available() else "cpu"model = VAE(data.shape[1], 8).to(device) optimizer = optim.Adam(model.parameters(), lr=1e-2200)
Il training:
train_loader = make_data_loader(data, batch_size=1024, drop_last=True, shuffle=True, device=device)epochs = 50train(model, train_loader, epochs, device) epochs = 50train(model, train_loader, epochs, device)
Ottenere le rappresentazioni latenti:
with torch.no_grad(): model.eval() data = LOAD DATA data = torch.from_numpy(data).float().to(device) em, _ = model.encode(data)
Visualizzare:
import randomsidx = random.sample(range(len(data)), 10000) # just use 10000 em_2d = umap.UMAP().fit_transform(em.cpu().numpy()[sidx])plt.figure(figsize=(10, 10)) sns.scatterplot(x=em_2d.T[0], y=em_2d.T[1], hue=truth[sidx]) plt.legend(bbox_to_anchor=(1.05, 1))
Binning nel caso di metagenomica
Una volta addestrato il VAE si ottengono le rappresentazioni latenti. In questo esempio ho utilizzato UMAP per proiettare un campione di 10000 reads in uno spazio 2D per poterlo visualizzare. Si presentava cosi:

Guardando la figura possiamo vedere gruppi di punti di dati qui e là. Si può facilmente utilizzare uno strumento come HDBSCAN per estrarre i cluster più densi.
Osservazioni finali
L’idea di utilizzare i VAE per fare binning è stata presentata da VAMB per assemblare binning. Tuttavia VAMB spesso richiede un numero elevato di contig (più di 10000 all’incirca). Questo perchè servono sempre più dati per ottenere risultati migliori in ambito deep learning. Considerando tutte queste sfide e opportunità, abbiamo sviluppato il nostro strumento per raggruppare reads di metagenomica, LRBinner. Qui abbiamo sempre milioni di reads. Lo strumento completo LRBinner, in realtà, è molto più complicato di quello che vi ho presentato in questo articolo. Tuttavia, l’intuizione e le idee rimangono le stesse. Abbiamo anche utilizzato un diverso algoritmo di clustering. Se siete interessati, date un’occhiata qua: https://github.com/anuradhawick/LRBinner/.
Trovate il Notebook Jupyter completo qui. Mentre il codice Pytorch originale è qui.
Spero che la lettura di questo articolo vi sia piaciuta. Buon proseguimento!
Share: