Scoperta di farmaci con reti neurali a grafo – parte 2
Scopri come prevedere la tossicità con i GNN utilizzando Deepchem, una libreria di deep learning per le scienze della vita.

Materiale correlato
Introduzione
In questo articolo, tratteremo un altro fattore cruciale che determina se il farmaco può superare i test di sicurezza: la tossicità. In effetti, la tossicità rappresenta il 30% dei farmaci candidati respinti, rendendolo uno dei fattori più importanti da considerare durante la fase di sviluppo del farmaco [1]. L’apprendimento automatico si rivelerà qui molto utile in quanto può filtrare i candidati farmaci tossici nella fase iniziale del processo di scoperta dei farmaci.
In questo articolo darò per scontato che avete già letto il mio articolo precedente che spiega alcuni termini e argomenti che utilizzerò anche in questo articolo 🙂 Iniziamo!
Affrontare il problema con le reti neurali a grafo
La parte relativa al feature engineering è molto simile a quella vista nella parte 1. Per convertire una struttura molecolare in un input per una GNN possiamo creare impronte digitali delle molecole o inserirle in una rete neurale a grafo utilizzando matrici di adiacenza e vettori delle feature. Queste feature possono essere generate automaticamente da software esterni come RDKit o Deepchem, per cui non è necessario preoccuparsene troppo.
Toxicity
La grande differenza sta nel task stesso di Machine Learning. Predirre la tossicità rappresenta un task di classificazione, al contrario della previsione di solubilità che è un task di regressione come possiamo ricordare dall’articolo precedente. Ci sono molti effetti tossici diversi come la cancerogenicità, tossicità respiratoria, irritazione/corrosione e altri [2]. Questo rende la sfida un poco più complicata in quanto dobbiamo gestire un dataset di classi sbilanciate.
Fortunatamente, i set di dati sulla tossicità sono spesso considerevolmente più grandi delle controparti di solubilità. Ad esempio, il set di dati Tox21 ha ~ 12k campioni di addestramento quando il set di dati Delaney utilizzato per la previsione della solubilità ha solo ~ 3k campioni di addestramento. Ciò rende le architetture delle reti neurali un approccio più promettente da utilizzare in quanto possono acquisire più informazioni nascoste.
Set di dati Tox21
Il set di dati Tox21 è stato creato come un progetto che sfida i ricercatori a sviluppare modelli di apprendimento automatico che raggiungano le massime prestazioni sui dati forniti. Contiene 12 etichette distinte e ognuna indica un diverso effetto di tossicità. Complessivamente, il set di dati ha 12.060 campioni di addestramento e 647 campioni di test.
L’approccio vincente per questa sfida è stato DeepTox [3] che è una pipeline di deep learning che utilizza descrittori chimici per prevedere le classi di tossicità. Suggerisce fortemente che il deep learning è l’approccio più efficace e che le reti neurali a grafo hanno il potenziale per ottenere prestazioni ancora più elevate.
Parte pratica con Deepchem
In primo luogo, importiamo le librerie necessarie. Niente di nuovo qui: useremo Deepchem per addestrare un modello GNN sui dati Tox21. Il GraphConvModel è un’architettura creata da Duvenaud, et al. Utilizza una versione modificata degli algoritmi delle impronte digitali per renderli differenziabili (quindi possiamo fare un aggiornamento del gradiente). È una delle prime architetture GNN progettate per gestire strutture molecolari come grafici.
Deepchem contiene una comoda API per caricare Tox21 per noi con . load_tox21 funzione. Scegliamo un featurizzatore come GraphConv: creerà descrittori chimici (cioè funzionalità) per soddisfare i requisiti di input per il nostro modello. Poiché si tratta di un’attività di classificazione, il punteggio ROC AUC verrà utilizzato come metrica.
La bellezza del Deepchem è che i modelli utilizzano API simili a Keras. Possiamo addestrare il modello con . funzione fit. Passiamo len(tox21_tasks) negli argomenti del modello, che è un numero di etichette (12 in questo caso). Questo imposterà la dimensione di output del livello finale su 12. Utilizziamo una dimensione del batch di 32 per accelerare il tempo di calcolo e specificare che il modello viene utilizzato per l’attività di classificazione. Il modello richiede diversi minuti per il training sui notebook di Google Colab.
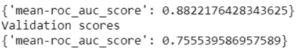
Una volta completato il training, possiamo valutare il modello. Niente di difficile anche qui: possiamo ancora usare l’API Keras per quella parte. I punteggi ROC AUC sono ottenuti con la funzione .evaluate.
Nel mio caso, il punteggio ROC AUC del treno era superiore al punteggio ROC AUC di convalida. Ciò potrebbe indicare che il modello è troppo adatto ad alcune molecole.
Puoi fare molto di più con Deepchem che. Contiene diversi modelli GNN facili da usare come in questo tutorial. Consiglio vivamente di guardare i loro tutorial. Per il compito di tossicità, hanno raccolto diversi esempi che funzionano con diversi modelli. Puoi trovarlo qui.
Grazie per aver letto l’articolo, spero che ti sia stato utile!
Su di me
Sono uno studente di MSc In Intelligenza Artificiale presso l’Università di Amsterdam. Nel mio tempo libero, puoi trovarmi a giocherellare con i dati o a eseguire il debug del mio modello di deep learning (giuro che ha funzionato!). Mi piace anche fare escursioni 🙂
Ecco i miei profili social, se vuoi rimanere in contatto con i miei ultimi articoli e altri contenuti utili:
Referenze
[1] “Can the pharmaceutical industry reduce attrition rates? “: https://pubmed.ncbi.nlm.nih.gov/15286737/
[2] “In Silico Prediction of Chemical Toxicity for Drug Design Using Machine Learning Methods and Structural Alerts”: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5826228/
[3] DeepTox: Toxicity Prediction using Deep Learning: https://www.frontiersin.org/articles/10.3389/fenvs.2015.00080/full
Share:


