DeepChem — Un framework per l’utilizzo di ML e DL per Life Science e Chemioinformatica
Democratizzare l'uso di ML e DL nella scoperta di farmaci, nella scienza dei materiali, nella chimica quantistica e nella biologia. Articolo di Aditya Bhattacharya

L’applicazione di Machine Learning e Deep Learning per la scoperta di farmaci, la genomica, la microscopia e la chimica quantistica può creare un impatto radicale e ha il potenziale per accelerare significativamente il processo di ricerca medica e sviluppo di vaccini, che è una necessità per qualsiasi pandemia come Covid19.
Prima ancora di iniziare, questo articolo è di altissimo livello e specificamente mirato per Data Scientist e ricercatori ML interessati alla scoperta di farmaci, specialmente durante il periodo di una pandemia esistente come Covid19. Se sei uno con un forte background in Bio-informatica o Chem-informatica e vuole avventurarsi nel mondo della scienza dei dati per questi casi d’uso, per favore contattami attraverso una delle opzioni menzionate qui, e possiamo discutere alcune opportunità interessanti per il bene superiore dell’umanità.
DeepChem, un framework open source, che utilizza internamente TensorFlow, che è stato specificamente progettato per semplificare la creazione di modelli di deep learning per varie applicazioni di scienze della vita.
In questo tutorial, vedremo come impostare DeepChem e vedremo come utilizzare DeepChem per:
- addestrare un modello in grado di prevedere la tossicità delle molecole
- addestramento di un modello per prevedere la solubilità delle molecole
- utilizzo di stringhe SMART per interrogare strutture molecolari.
Configurazione di DeepChem
Anche se, in più fonti, ho visto che gli utenti hanno espresso la loro preoccupazione nella configurazione di DeepChem in ambienti Windows, Linux e Mac, ma ho trovato abbastanza facile farlo usando pip installer.
Il team di sviluppo di DeepChem è molto attivo e fornisce build giornaliere, quindi vorrei che tutti dessero un’occhiata alla loro pagina pypi: https://pypi.org/project/deepchem/#history e installare una versione adatta nel caso in cui l’ultima versione abbia qualche problema. Una semplice installazione di pip deepchem installerebbe l’ultima versione.
Successivamente, insieme a DeepChem, è necessario installare TensorFlow. Avevo installato l’ultima versione di TensorFlow utilizzando pip install tensorflow e RDkit che è un pacchetto software Cheminformatics open source. Per RDkit e per l’installazione in Windows, non ho trovato alcun programma di installazione pip affidabile, quindi l’ho installato da https://anaconda.org/rdkit/rdkit utilizzando il programma di installazione conda: conda install -c rdkit rdkit
Una volta installati questi tre moduli, siamo pronti per iniziare con i nostri esperimenti.
Prevedere la tossicità delle molecole
La tossicità molecolare può essere definita come la somma degli effetti nocivi esibiti da una sostanza su qualsiasi organismo. I metodi computazionali possono effettivamente determinare la tossicità di un dato composto utilizzando proprietà chimiche e strcutrali della molecola e caratterizzazione molecolare utilizzando descrittori molecolari (Dong et al., 2015) e impronte digitali (Xue e Bajorath, 2000), possono estrarre efficacemente le informazioni chimiche e strutturali inerenti a una data molecola per approcci basati sulla previsione.
Per prevedere la tossicità, utilizzeremo il set di dati sulla tossicità Tox21 di MoleculeNet e useremo DeepChem per caricare il set di dati richiesto.
import numpy as np import deepchem as dc tox21_tasks, tox21_datasets, transformers = dc.molnet.load_tox21()
Dopo questo vedremo tutte le classi di tossicità, basta stampare tox21_tasks
['NR-AR', 'NR-AR-LBD', 'NR-AhR', 'NR-Aromatase', 'NR-ER', 'NR-ER-LBD', 'NR-PPAR-gamma', 'SR-ARE', 'SR-ATAD5', 'SR-HSE', 'SR-MMP', 'SR-p53']
Possiamo dividere l’intero set di dati in set di dati di addestramento, test e convalida per:
train_dataset, valid_dataset, test_dataset = tox21_datasets
Se controlliamo la distribuzione del set di dati, vedremo che il set di dati non è bilanciato, quindi dovremmo bilanciare il set di dati poiché in genere stiamo cercando di risolvere un problema di classificazione MultiClass. E quindi se il set di dati non è bilanciato, la classe di maggioranza aggiungerà pregiudizi al classificatore, che distorcerà i risultati. Quindi, l’oggetto trasformatore utilizzato per impostazione predefinita è un trasformatore di bilanciamento.
print(transformers) [<deepchem.trans.transformers.BalancingTransformer at 0x26b5642dc88>]
ora, per la parte di formazione :
model = dc.models.MultitaskClassifier(n_tasks=12, n_features=1024, layer_sizes=[1000]) model.fit(train_dataset, nb_epoch=10) metric = dc.metrics.Metric(dc.metrics.roc_auc_score, np.mean) train_scores = model.evaluate(train_dataset, [metric], transformers) test_scores = model.evaluate(test_dataset, [metric], transformers)
Ora, il sottomodulo di DeepChem contiene una varietà di dc.models diversi modelli specifici per le scienze della vita.
E infine vediamo, che i punteggi finali AUC-ROC sono:
{'training mean-roc_auc_score': 0.9556297601807405}
{'testing mean-roc_auc_score': 0.7802496964641786}
Questo ci mostra che c’è un certo over-fitting nel modello in quanto i punteggi delle metriche del set di dati di test sono molto inferiori rispetto al set di treni. Ma, tuttavia, ora abbiamo un modello in grado di prevedere la tossicità delle molecole!
Prevedere la solubilità delle molecole
La solubilità è una misura, che mostra quanto facilmente una molecola può dissolversi in acqua. Per qualsiasi scoperta di farmaci, è molto importante controllare la solubilità del composto in quanto il farmaco dovrebbe dissolversi nel flusso sanguigno del paziente per avere l’effetto terapeutico richiesto. Di solito, i chimici medicinali trascorrono molto tempo a modificare le molecole per aumentare questa proprietà di solubilità. In questa sezione useremo DeepChem per prevedere la solubilità delle molecole.
Utilizzeremo il set di dati delaney di MoleculeNet, disponibile anche in DeepChem, per prevedere la solubilità molecolare.
# load the featurized data tasks, datasets, transformers = dc.molnet.load_delaney(featurizer='GraphConv')# Split into traintest-validation dataset train_dataset, valid_dataset, test_dataset = datasets# Fit the model model = dc.models.GraphConvModel(n_tasks=1, mode='regression', dropout=0.2) model.fit(train_dataset, nb_epoch=100)# Use r2 score as model evaluation metric metric = dc.metrics.Metric(dc.metrics.pearson_r2_score) print(model.evaluate(train_dataset, [metric], transformers)) print(model.evaluate(test_dataset, [metric], transformers))
Anche nel primo passaggio, vediamo alcuni overfitting dai risultati della valutazione del modello.
{'training pearson_r2_score': 0.9203419837932797}
{'testing pearson_r2_score': 0.7529095508565846}
Vediamo come prevedere la solubilità per un nuovo insieme di molecole:
smiles = ['COC(C)(C)CCCC(C)CC=CC(C)=CC(=O)OC(C)C', 'CCOC(=O)CC', 'CSc1nc(NC(C)C)nc(NC(C)C)n1', 'CC(C#C)N(C)C(=O)Nc1ccc(Cl)cc1', 'Cc1cc2ccccc2cc1C']
Successivamente, dobbiamo presentare questi nuovi set di molecole dal loro formato SMILES
from rdkit import Chem mols = [Chem.MolFromSmiles(s) for s in smiles] featurizer = dc.feat.ConvMolFeaturizer() x = featurizer.featurize(mols)predicted_solubility = model.predict_on_batch(x) predicted_solubility
E così, possiamo vedere i valori di solubilità previsti :
array([[-0.45654652], [ 1.5316172 ], [ 0.19090167], [ 0.44833142], [-0.32875094]], dtype=float32)
Abbiamo visto molto facilmente come DeepChem rende molto facile per i due casi d’uso di cui sopra, che potrebbero richiedere molto tempo per un chimico umano per risolvere questi problemi!
Per la parte finale, vedremo alcune visualizzazioni e tecniche di query disponibili come parte di RDkit, che è molto richiesto quando qualcuno sta lavorando per tali casi d’uso.
Stringhe SMART per interrogare le strutture molecolari
SMARTS è un’estensione del linguaggio SMILES descritto in precedenza che può essere utilizzata per creare query.
# To gain a visual understanding of compounds in our dataset, let's draw them using rdkit. We define a couple of helper functions to get startedimport tempfile from rdkit import Chem from rdkit.Chem import Draw from itertools import islice from IPython.display import Image, displaydef display_images(filenames): """Helper to pretty-print images.""" for file in filenames: display(Image(file))def mols_to_pngs(mols, basename="test"): """Helper to write RDKit mols to png files.""" filenames = [] for i, mol in enumerate(mols): filename = "%s%d.png" % (basename, i) Draw.MolToFile(mol, filename) filenames.append(filename) return filenames
Ora, prendiamo una stringa SMILES di esempio e visualizziamo la struttura molecolare.
from rdkit import Chem from rdkit.Chem.Draw import MolsToGridImage smiles_list = ["CCCCC","CCOCC","CCNCC","CCSCC"] mol_list = [Chem.MolFromSmiles(x) for x in smiles_list] display_images(mols_to_pngs(mol_list))
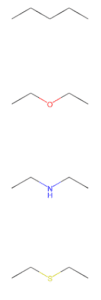
Questo è il modo in cui le strutture visive sono formate dalla stringa SMILES.
Ora, supponiamo di voler interrogare la stringa SMILES che ha tre carboni adiacenti.
query = Chem.MolFromSmarts("CCC")
match_list = [mol.GetSubstructMatch(query) for mol in
mol_list]
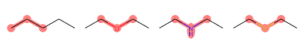
MolsToGridImage(mols=mol_list, molsPerRow=4,
highlightAtomLists=match_list)
![]()
Vediamo che la parte evidenziata rappresenta il composto con tre carboni adiacenti.
Allo stesso modo, vediamo alcune query di caratteri jolly e altre opzioni di query di sottostruttura.
query = Chem.MolFromSmarts("C*C")
match_list = [mol.GetSubstructMatch(query) for mol in
mol_list]
MolsToGridImage(mols=mol_list, molsPerRow=4,
highlightAtomLists=match_list)
query = Chem.MolFromSmarts("C[C,N,O]C")
match_list = [mol.GetSubstructMatch(query) for mol in
mol_list]
MolsToGridImage(mols=mol_list, molsPerRow=4,
highlightAtomLists=match_list)
![]()
Pertanto, possiamo vedere che anche la sottoquery selettiva può essere facilmente gestita.
Quindi, questo ci porta alla fine di questo articolo. So che questo articolo era di altissimo livello e specificamente mirato per Data Scientist e ricercatori ML interessati alla scoperta di farmaci, specialmente durante il periodo di una pandemia esistente come Covid19. Spero di essere stato in grado di aiutarti! Se sei una persona con un forte background in Bio-informatica o Chimica-informatica e vuoi avventurarti nel mondo della scienza dei dati, ti prego di contattarmi attraverso una delle opzioni menzionate qui. Continua a seguire: https://medium.com/@adib0073 e il mio sito web: https://www.aditya-bhattacharya.net/ per saperne di più!
Se, invece, siete interessati ad un corso che vi permetta di approfondire l’applicazione della Data Science in ambito biologico iscrivetevi al corso “Data Science e Bioinformatica” erogato da Francesco Lescai esclusivamente per Deep Learning Italia!
Share:


